Micro-ARN
El micro-ARN o mi-ARN es un ARN monocatenario, de una longitud de entre 21 y 25 nucleótidos, que tiene la capacidad de regular la expresión de otros genes mediante diversos procesos, utilizando para ello la ruta de ribointerferencia.[1][2][3]

Fueron descritos inicialmente en 1993 por Rosalind Lee y colaboradores en el laboratorio de Víctor Ambros,[4] sin embargo, el término «micro-ARN» solo se acuñó en 2001 en un conjunto de tres artículos publicados en Science (26 de octubre de 2001).[5] A principios de 2008, análisis computacionales realizados por IBM sugerían la presencia de alrededor de 50,000 micro-ARN diferentes en el genoma humano, cada uno tal vez con alrededor de miles de micro-ARN dianas potenciales.[6]
Definición
editarUna vez descubiertos los siARN y la existencia en las células de proteínas que catalizan la degradación del mARN, los investigadores se preguntaron si los siARN también estaban codificados en el genoma, y empezaron a purificar pequeños ARN (19-25 nt) a partir de diferentes especies animales. Sin embargo, no encontraron siARN, sino los denominados micro-ARN, que se habían identificado anteriormente de forma independiente.[4]
Los micro-ARN son moléculas de ARN transcritas a partir de genes de ADN,[1] pero no son traducidas a proteínas. Se expresan en una amplia variedad de seres vivos, desde plantas hasta gusanos y seres humanos. Muchos micro-ARN están bien conservados entre especies,[8] y muchos componentes de la maquinaria de los micro-ARN se han encontrado incluso en arqueas y bacterias, lo que revela que su origen es muy antiguo. Algunos recuentos de micro-ARN en humanos identificaban hasta 800, lo que implicaría que los micro-ARN podrían representar como mínimo el 3 por ciento de los genes humanos.[9]
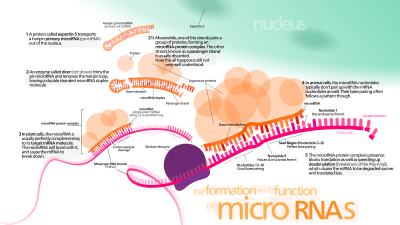
La secuencia de ADN que codifica para un gen de micro-ARN tiene una longitud que supera al tamaño final del propio micro-ARN e incluye la región micro-ARN y una región que es complementaria a la anterior, lo que permite su apareamiento. Esto conlleva que, durante la transcripción de esta secuencia de ADN, se forman regiones que tienen la capacidad de formar una horquilla y generar un ARN bicatenario primario largo conocido como pri-micro-ARN. Posteriormente, una enzima nuclear llamada DROSHA corta las bases de la horquilla, formando lo que se denomina pre-micro-ARN. Este pre-micro-ARN es transportado desde el núcleo al citoplasma por la exportina 5. Una vez que el pre-micro-ARN está en el citoplasma es fragmentado por la enzima DICER, que lo corta hasta la longitud final de 20-25 nucleótidos.[7]
Función
editarLa función de los micro-ARN está relacionada con la regulación de la expresión génica.[1] De esta forma un micro-ARN es complementario de una parte de uno o más ARN mensajeros (ARNm). Los micro-ARN de animales suelen mostrar complementariedad imperfecta con la región 3' UTR, y generalmente inhiben la traducción del mARN, mientras que los de plantas suelen mostrar complementariedad perfecta con regiones codificantes e inducen el corte y la posterior degradación del mARN diana (como ocurre con los siARNs en animales).[7]
Antes de clasificarlos como parte de la ruta del iARN, los micro-ARN fueron identificados inicialmente en gusanos, en los cuales regulan las fases del desarrollo,[10] pero en la actualidad se sabe que están implicados en una amplia variedad de procesos del desarrollo y que además podrían tener una función en el establecimiento de redes y en el ajuste fino de la expresión génica en la célula.[11][8] Dado que el número de dianas potenciales de los micro-ARN aumenta al número de miles (alrededor del 30 % de los genes humanos), los micro-ARN podrían constituir otra capa del circuito regulatorio que existe en las células.[12] Según esto, cualquier desregulación de los micro-ARN podría conllevar grandes problemas regulatorios en la célula, induciendo quizá fenotipos cancerosos. De hecho, se ha mostrado que los perfiles de expresión de los micro-ARN están modificados en un gran número de tipos de cáncer[13] y que la sobreexpresión forzada de los micro-ARN podría conducir al desarrollo de tumores.[14]
Características de los micro-ARN
editarLos micro-ARN se transcriben a partir de diferentes localizaciones genómicas como largos tránscritos primarios (pri-micro-ARN) por la ARN-polimerasa II.[15]
Los micro-ARN pueden encontrarse en muchos tipos de loci en el genoma:[16]
- La transcripción de los micro-ARN puede estar regulada independientemente por promotores específicos, como ocurre con miR-1–1 y miR-133a-2, que están regulados por los factores de transcripción factores de respuesta al suero y MyoD[17] (ver el apartado Funciones durante el desarrollo para más detalles). Tales micro-ARN pueden de todas maneras estar localizados en intrones pero a menudo con una orientación antisentido.
- La mayoría de los precursores de micro-ARN presentes en intrones tienen la misma orientación que el gen en el que se alojan y son inicialmente transcritos como parte de su ARN precursor.[8] Por ejemplo, miR-208, que se expresa específicamente en el corazón de humano y ratón, reside en el intrón 28 de la cadena pesada de la α-miosina cardiaca. Los pri-micro-ARN intrónicos pueden producirse por corte del intrón por la endonucleasa DROSHA, después de que se haya realizado el proceso de ayuste.
- Con pocas excepciones, los micro-ARN que están integrados o se superponen con exones de tránscritos conocidos, están siempre en la misma orientación, y la mayoría se encuentran en las regiones no-codificantes UTR 5′ o 3′ (por ejemplo, miR-198 en follistatin-like 1).
Aproximadamente el 50 % de los micro-ARN están en clusters (grupos) de micro-ARN que están inicialmente codificados como un tránscrito policistrónico (que incluye varios genes),[18] que posteriormente se fragmenta en múltiples micro-ARN. En la mayoría de los casos, los micro-ARN policistrónicos comparten el mismo patrón de expresión. Sin embargo, los niveles relativos de los micro-ARN dentro del grupo parecen estar regulados de una manera dependiente del desarrollo y la homeostasis, lo que sugiere una complejidad aún no definida en la regulación de la expresión génica.
Biogénesis y procesamiento
editarLos genes que codifican micro-ARN son mucho más largos que los micro-ARN procesados maduros; los micro-ARN se transcriben inicialmente como tránscritos primarios o pri-micro-ARN con una caperuza en 5' y una cola de poli-adeninas (poly-A) en 3' y se procesan en el núcleo celular en estructuras cortas de 70-nucleótidos en forma de tallo-asa (stem-loop) conocidas como pre-micro-ARN. En animales este procesamiento se realiza por un complejo proteico llamado Microprocesador, que consta de la nucleasa DROSHA[19] y la proteína de unión a ARN de doble hélice, PASHA.[20][21] Estos pre-micro-ARN son luego procesados a micro-ARN maduros en el citoplasma mediante la interacción con la ribonucleasa DICER, que también inicia la formación del complejo RISC (ARN-induced silencing complex).[22] Este complejo es el responsable del silenciamiento de genes que se observa debido a la expresión de los micro-ARN y a la interferencia de ARN mediada por siARNs. La ruta varía ligeramente en plantas, debido a que carecen de homólogos de DROSHA; en su lugar, sólo homólogos de DICER realizan algunos de los pasos del procesamiento.[23] La ruta también es diferente para los micro-ARN derivados de tallos-asas (stem-loops) procedentes de secuencias intrónicas; en este caso se procesan por DICER pero no por DROSHA.[24] Tanto la hebra sentido como la antisentido del ADN pueden funcionar como molde para producir micro-ARN.[25]
DROSHA[26] (ARNSEN en humanos)[19] es una proteína nuclear de un tamaño entre 130 y 160 kDa (kiloDalton). Contiene los dominios siguientes:
- dos dominios ARNasa III (los dominios catalíticos)
- un dominio dsRBD (double-strand ARN-binding domain, de unión al ARN doble hebra)
- dominios conservados (en el extremo N-terminal), de los que no se conoce la función.
DROSHA funciona en un complejo (Microprocesador), conjuntamente con una proteína de unión a ARN (denominada PASHA en Drosophila o DGCR8 en mamíferos). DGCR8[27][28] es una proteína de unión a ADN que reconoce la zona de unión dsARN–ssARN (ARN doble hebra-simple hebra) y posiciona a la ribonucleasa DROSHA a una distancia de 11 nucleótidos (que corresponde a una vuelta de hélice) desde la zona de unión. Por tanto, la función de DGCR8 en el complejo Microprocesador es análoga al dominio PAZ de DICER; DGCR8 proporciona especificidad de sustrato y posiciona adecuadamente el centro de la ribonucleasa DROSHA. Sin embargo, en el caso de DROSHA-DGCR8, el dominio de especificidad está localizado en una cadena polipeptídica separada de los dominios ARNasa III. Se ha propuesto que el dominio WW de unión a prolinas de DGCR8 interacciona con el extremo N-terminal de DROSHA, rico en prolinas. Es posible que DROSHA tenga otras posibles proteínas asociadas que presenten dominios WW que aporten especificidades de sustrato alteARNtivas y funciones biológicas adicionales de DROSHA.
El procesamiento eficiente de los pre-micro-ARN por DROSHA requiere la presencia de largas colas de ARN de hebra simple tanto en el extremo 3' como 5' de la molécula en horquilla.[29] Estos motivos de ARN de hebra simple (ssARN) pueden variar en composición, mientras que su longitud es de gran importancia para que el procesamiento tenga lugar. Un análisis bioinformático de pri-micro-ARN en humanos y moscas identificó regiones estructurales similares, denominadas 'segmentos basales (basal segments)', 'tallos inferiores (lower stems)', 'tallos superiores (upper stems)' y 'asas terminales (terminal loops)'; basándose en estas estructuras conservadas, se han determinado perfiles termodinámicos de los pri-micro-ARN.[30] El complejo de DROSHA (Microprocesador) corta la molécula de ARN ~2 vueltas de hélice a partir del asa terminal y ~1 vuelta de hélice a partir de los segmentos basales. En la mayoría de las moléculas analizadas esta región contiene nucleótidos no apareados y la energía libre del dúplex es relativamente alta en comparación con las regiones tallo superior e inferior [cita requerida]. La mayor parte de los pre-micro-ARN no presentan una estructura de ARN doble hebra (dsARN) perfecta con un asa terminal final. Existen algunas explicaciones posibles para esta selectividad. Una podría ser que dsARN más largos de 21 pares de bases activan la respuesta de Interferón y la maquinaria antiviral de la célula. Otra explicación plausible podría ser que el perfil termodinámico del pre-micro-ARN determina qué hebra será incorporada en el complejo DICER. De hecho, se han detectado claras similitudes entre pri-micro-ARN codificados bien en hebras 5' o 3'.[30]
Una vez generado el pre-micro-ARN, DICER corta el tallo-asa (stem-loop) y se forman dos moléculas complementarias cortas, pero solo una se integra en el complejo RISC (la antisentido), como ocurre con los siARNs. Esta hebra se conoce como la hebra guía, que es seleccionada por la proteína Argonauta, la ARNasa catalíticamente activa en el complejo RISC, en función de la estabilidad del extremo 5'.[31] La otra hebra (la sentido), conocida como anti-guía o hebra pasajera, es degradada por el complejo RISC.[32] Después de su integración en el complejo RISC, ahora activado (ver las notas sobre el complejo RISC en siARNs), los micro-ARN se emparejan de acuerdo con su secuencia de bases con la molécula de ARNm complementaria, y en animales, a diferencia con los siARN, en la mayoría de los casos inducen la inhibición de la traducción de dicho mARN.
Sin embargo, a pesar de que DICER es una enzima fundamental en el procesamiento de los micro-ARN, se ha identificado una ruta de biogénesis de micro-ARN independiente de DICER que utiliza la actividad catalítica de corte de Argonauta2 (Ago2). Así, a diferencia de otros micro-ARN, los niveles de algunos micro-ARN (miR-451-5', miR-2190-5', miR-2190-3', y miR-735-5') no se alteran en mutantes con pérdida de función de DICER, pero disminuyen en mutantes MZago2 (cigótico mateARNl). En el caso de miR-451 (un micro-ARN implicado en la diferenciación de la línea eritroide[33]), el procesamiento de pre-miR-451 requiere la actividad catalítica de Ago2 in vivo. Los mutantes MZago2 muestran un retraso en la eritropoyesis que puede rescatarse utilizando Ago2 de tipo salvaje o con dúplex de miR-451, pero no con Ago2 catalíticamente inactiva. Por ello, se ha sugerido que Ago2 es capaz de generar micro-ARN funcionales independientemente de Dicer.[34] Resultados similares se han observado en organismos diferentes.[35]
Como se indica en el caso de los siARN, todavía no está claro cómo el complejo RISC activado localiza los mARN complementarios en el interior celular. Las proteínas Argonauta, los componentes catalíticos de RISC, están localizadas en regiones específicas del citoplasma denominadas P-bodies (cuerpos-P, o cuerpos citoplásmicos o cuerpos GW, porque contienen la proteína GW182), los cuales son regiones con altas tasas de degradación de mARN;[36] también se ha detectado actividad micro-ARN en los P-bodies.[37] La alteración de los P-bodies disminuye la eficiencia del proceso de iARN, lo que sugeriría que los P-bodies podrían ser un lugar crítico para el proceso de iARN.[38] Sin embargo, estudios posteriores han demostrado que los P-bodies no son imprescindibles para el proceso de iARN, ya que células sin P-bodies pueden producir silenciamiento tanto con siARN como con micro-ARN.[39]
Modo de funcionamiento
editarA pesar del importante progreso realizado en la comprensión de la biogénesis y la función de los micro-ARN, los mecanismos utilizados por los micro-ARN para regular la expresión génica permanecen bajo un intenso debate.[40] En efecto, existen trabajos publicados que indican que los micro-ARN en células animales reprimen la expresión génica de cuatro formas diferentes:
- degradación de la proteína durante la traducción
- inhibición de la elongación de la traducción
- terminación prematura de la traducción (disgregación de los ribosomas)
- inhibición de la iniciación de la traducción
Además, los micro-ARN en animales pueden inducir una degradación significativa de los mARN diana (como los micro-ARN de plantas), a pesar del apareamiento imperfecto mARN-micro-ARN. Sin embargo, el mecanismo de degradación suele ser diferente: los micro-ARN inducen la degradación de los mARN diana mediante la eliminación de la caperuza (en el extremo 5') y de la cola de poliadeninas (poly-A, en el extremo 3').[41] Finalmente, los miARN podrían también silenciar sus mARN dianas secuestrándolos en loci (sitios) citoplásmicos discretos, los cuerpos de procesamiento de mARN o P bodies, que carecen de maquinaria de traducción. Sin embargo, a pesar de las discrepancias existentes entre los diferentes mecanismos propuestos, los apoyos experimentales para cada mecanismo son variados, y son el objeto actual de intensos estudios, para tratar de elucidarlas. Se ha sugerido que las diferencias observadas se deben a deficiencias en los experimentos realizados, en algunos casos originadas por la utilización de modelos erróneos en los estudios de regulación de la traducción.[42]
Por último, en estudios recientes se ha detectado que en determinadas condiciones, los micro-ARN pueden también activar la síntesis proteica.[43]
Características generales de los micro-ARN:[40]
- Se asocian a la región 3’ UTR de los ARNm diana.
- Hacen falta muchos sitios de unión para activar la respuesta de los micro-ARN (la unión de uno solo no produce efectos significativos).
- Un mARN puede estar regulado por diferentes micro-ARN.
- Un único micro-ARN puede controlar la actividad de cientos de mARN diferentes.[44] Los primeros estudios a gran escala publicados en Nature en 2008, que utilizan una variante de la técnica de espectrometría de masas denominada SILAC (marcado estable de aminoácidos con isótopos pesados en cultivo celular, Stable Isotope Labelling with Amino acids in Cell culture) para detectar las proteínas afectadas al reducir o aumentar los niveles de un micro-ARN concreto, muestran que en efecto, un único micro-ARN puede reducir los niveles de cientos de proteínas mediante el bloqueo de su traducción, no sólo degradando sus mARN.[45][46] El descubrimiento más llamativo de estos estudios es que los efectos de los micro-ARN sobre las proteínas son habitualmente bastante modestos, modificando sus niveles de expresión tan sólo en un factor 2. Sin embargo, a pesar de que los efectos de los micro-ARN pueden ser sutiles, también pueden ser potentes: los micro-ARN parecen intervenir en la regulación de la expresión génica realizando un ajuste fino, de forma compleja e interconectada.
- La especificidad y la función de los micro-ARN están determinados por los nucleótidos 2 a 7 de la parte 5' de los micro-ARN maduros (la llamada región «semilla» del micro-ARN): dichos nucleótidos deben ser obligatoriamente complementarios al mARN diana.[47]
- Un micro-ARN puede ser funcional aunque no haya sido sintetizado en el núcleo: un micro-ARN introducido en la célula por transfección puede inhibir eficazmente la síntesis de proteínas. En este caso, como ocurre con los siARNs, el efecto del micro-ARN se puede modular extracelularmente utilizando moléculas químicas pequeñas. En un estudio publicado en agosto de 2008, se desarrolló un ensayo basado en células para monitorizar la actividad de la ruta del iARN, y los autores detectaron que la molécula enoxacin (Penetrex) mejora la degradación del mARN mediada por siARN, y promueve la biogénesis de los micro-ARN endógenos.[48] Este artículo prueba que pequeñas moléculas químicas pueden mejorar la eficacia de la ruta del iARN, lo que podría ser útil en el desarrollo de herramientas terapéuticas y para la investigación.
- El micro-ARN es el responsable de la especificiad de sustrato (el mARN diana) del miRNP (complejo micro-ribo-nucleo-proteico, el micro-ARN unido al complejo RISC).
Función biológica
editarFunción ante la infección vírica y la respuesta inmunitaria
editar- Función anti-viral: el micro-ARN miR-32 tiene como diana una secuencia presente en la región no traducida del extremo 3’ de todos los mARN retrovirales[49]
- Función pro-viral: el micro-ARN miR-122, que se expresa específicamente en el hígado, es necesario para que el virus de la hepatitis C se exprese de manera eficiente.[50]
- Muchos micro-ARN que están regulados diferencialmente en líneas celulares hematopoyéticas tienen funciones importantes en la regulación del desarrollo y la función de las células del sistema inmunitario, y en las interacciones huésped-patógeno.[51]
Función en cáncer
editarLos micro-ARN pueden funcionar como supresores de tumores o como oncogenes; queda por demostrar su influencia concreta en cada tipo de cáncer.[52] De hecho, un estudio mostró que cerca del 50 % de los micro-ARN anotados en el ser humano están localizados en áreas del genoma conocidas como sitios frágiles,[53] que están asociadas con el desarrollo de cáncer.
La expresión de ciertos micro-ARN está correlacionada con varios tipos de cáncer, por lo que funcionarían como oncogenes. Por ejemplo, un informe de Sonoki y colaboradores[54] relacionó el gen mir-125b-1 con leucemia, y describió un paciente con leucemia linfoblástica aguda de precursores de células B que portaba una inserción del pre-micro-ARN en el locus de la cadena pesada de la inmunoglobulina. Aunque los investigadores no pudieron determinar cómo se modulaba la expresión de mir-125b-1 en las células tumorales, este estudio apoya el papel de este gen como un oncomir.
La primera indicación de que los micro-ARN podrían funcionar como supresores de tumores proceden de un informe de Calin y colaboradores[55] que mostraba que pacientes diagnosticados con una forma frecuente de leucemia en adultos (leucemia linfoide crónica de las células B o LLC), presentan a menudo deleciones o sub-regulación (downregulation) de dos genes de micro-ARN presentes en un clúster, mir-15a y mir-16-1. Deleciones dentro del locus 13q14 ocurren en más del 65 % de los casos de LLC, y en más del 50 % de los linfomas de las células del manto, el 16–40 % de los mielomas múltiples y el 60 % de los cánceres de próstata. Por tanto, se predijo que un gen supresor de tumores debía residir en esta región de 30-kb. Es interesante notar que mir-15a y mir-16-1 mapean dentro del intrón de un gen de ARNnc (ARN no codificante) para proteína, de función desconocida, denominado LEU2. Por otro lado, algunos estudios establecen una conexión entre la reducción de la expresión de let-7 (que regula la proliferación y diferenciación celular en C. elegans) y el incremento de la tumorigénesis y el pronóstico grave de los pacientes afectados.[56] Además, durante el desarrollo normal, LIN28 (un promotor de pluripotencia) puede prevenir la acumulación de let-7. De acuerdo con estos resultados, se ha propuesto que let-7 regularía la troncalidad (stemness) de las células, reprimiendo la auto-renovación y promoviendo la diferenciación, tanto durante el desarrollo normal como en cáncer.
Por otro lado, se ha observado además que los micro-ARN contribuyen a la progresión maligna del cáncer, específicamente mediando la invasión tumoral y la formación de metástasis.[57]
Función durante el desarrollo
editarPor ejemplo, en C. elegans, los micro-ARN permiten un paso rápido a través de las diferentes fases del desarrollo:[58] los micro-ARN lin-4[59] y let-7[60] controlan el momento en los que se define el destino de las células neuronales e hipodérmicas durante el desarrollo larvario.[61] lin-4, let-7 y otros genes de micro-ARN están conservados en mamíferos, y su función potencial en el desarrollo embrionario de mamíferos están bajo estudio activo. En C. elegans, los niveles de expresión de LIN-14[62] y LIN-28[63] disminuyen debido a la expresión del ARN de lin-4 al final del primer estadio larvario, para permitir la progresión a estadios larvarios posteriores. En los estadios larvarios finales, la expresión de LIN-41[64] y otros genes podría verse reducida por la expresión del ARN de let-7, liberando la represión de la expresión de la proteína LIN-29[65] y permitiendo la progresión al estadio adulto. Dado que el ARNm de lin-29 no posee sitios complementarios al ARN de let-7, probablemente lin-29 no es una diana directa de let-7.
Por otro lado, en miocitos de mamíferos, la activación del factor de transcripción SRF[66] induce la expresión de miR-1–1[67] y miR-1–2,[68] que a su vez reprime la expresión del factor de transcripción Hand2[69] y el ligando de Notch,[70] Delta, afectando por tanto la expansión de las células progenitoras o su diferenciación.[71] SRF también induce la expresión de miR-133a-1[72] y miR-133a-2,[73] lo que inhibe a su vez SRF en un lazo de retroalimentación (feedback loop). En músculo opera una ruta similar, excepto que la inhibición por retroalimentación de Mef2[74] y MyoD[75] ocurre cuando la expresión de HDAC4[76] está reducida debido al silenciamiento ejercido por miR-1–1 y miR-1–2.
Uno de los primeros micro-ARN detectados con una función en el desarrollo del sistema inmunitario[51] fue miR-181a;[77] este micro-ARN se expresa en altos niveles en las células del timo y en niveles menores en las células del corazón, los nódulos linfáticos y la médula ósea. En la médula ósea, miR-181a se expresa en niveles mayores por células B B220+ B que por células T CD3+. Específicamente, la expresión de miR-181a en células B derivadas de la médula ósea disminuye durante la maduración de las células B desde el estadio del desarrollo de pro-célula-B al estadio pre-célula-B. Además, la expresión ectópica de miR-181a en células enriquecidas en células madre y progenitoras hematopoyéticas resultó en un incremento en el porcentaje de células B CD19+ y un descenso en el porcentaje de células T CD8+ en ensayos de reconstitución de la médula ósea a corto plazo en ratones, demostrando que la especificidad de líneas celulares de los micro-ARN podría tener una función en la regulación del desarrollo de los linfocitos.
Por otro lado, también se ha detectado una distribución espacial de los micro-ARN: en embriones del pez cebra (zebrafish), los patrones de localización de micro-ARN individuales indican que su actividad podría estar limitada a los tejidos y órganos en los que se expresan. Por ejemplo, miR-206[78] se expresa sobre todo en el músculo, miR-126[79] en los vasos sanguíneos y el corazón, miR-200a[80] en el sistema de la línea lateral (un sistema mecanosensorial que detecta el movimiento del agua) y órganos sensoriales, y miR-30c[81] en el precursor de los riñones.[82]
Nuevas funciones mateARNs
editarDos estudios independientes en 2007 en ratones[83][84] indican que una cantidad significativa de micro-ARN maternos se heredan por los zigotos, y que dichos micro-ARN maternos podrían tener un papel importante en las etapas tempranas del desarrollo embrionario (efecto materno).
Homeostasis de colesterol y triglicéridos
editarEstudios de asociación de genoma completo (GWAS) se han empleado para identificar ciertos micro-ARNs que modulando la traducción del mARN funcionan como reguladores cruciales del colesterol, triglicéridos y homeostasis energética en el organismo.
Los micro-ARNs implicados en control metabólico y asociados a trastornos cardiometabólicos son: miR-128-1, miR-148a, miR-130b y miR-301b. Siendo los dos primeros los más relevantes en esta acción, ya que la introducción de precursores de estos micro-ARNs en células hepáticas humanas desencadenó una menor expresión del gen para el receptor de LDL y ABCA1 (ATP-binding cassette A1). Por el contrario, con la introducción de anti-micro-ARNs de estos, aumentaba la expresión del receptor de LDL y ABCA1.
El receptor de LDL capta LDL-colesterol, tiene papel, por tanto, en la depuración hepática y en la prevención de aterosclerosis y enfermedades cardiovasculares. Y estos micro-ARNs afectan a su expresión, disminuyéndola.
ABCA1 es un componente crítico en la ruta del colesterol total, ya que facilita la producción de HDL de novo en hígado e intestino, el cual es el responsable de eliminar el colesterol de las células periféricas y transportarlo al hígado para su excreción. estos micro-ARNs afectan a su expresión, y por tanto, a la salida de colesterol de las células para ser excretado. por el contrario, anti-micro-ARNs de estos, favorecen la expresión de ABCA1, produciendo mayores cantidades de HDL.
La sobreexpresión miR-128-1 y miR-148a principalmente, reduce fuertemente los niveles hepáticos de ABCA1 y LDL-R. También, miR 128-1 altera la expresión de IRS1 (receptor de insulina) contribuyendo a la resistencia a insulina, y micro-ARN-148a altera la expresión de CPT1A. El perfil de colesterol y lípidos circulantes en ratones que sobreexpresan estos micro-ARNs reveló una marcada reducción de los niveles plasmáticos de HDL-C en comparación con los controles. Debido a que se necesita ABCA1 para su síntesis, y este ha disminuido con la introducción de los micro-ARNs. Todo esto se revierte al introducir anti-micro-ARNs, por tanto, estos podrían ser aplicados en el tratamiento de enfermedades relacionadas con la homeostasis, sensibilidad glucídica, señalización de la insulina, triglicéridos y colesterol en sangre.
Véase también
editarReferencias
editar- ↑ a b c «micro-ARN». Real Academia Nacional de Medicina de España.
- ↑ Pillai, Ramesh S. (Diciembre de 2005). «MicroRNA function: multiple mechanisms for a tiny RNA?». RNA (New York, N.Y.) 11 (12): 1753-1761. ISSN 1355-8382. PMC 1370863. PMID 16314451. doi:10.1261/rna.2248605. Consultado el 23 de agosto de 2024.
- ↑ miRNAs, Human Health and Diseases.
- ↑ a b Lee, R. C.; Feinbaum, R. L.; Ambros, V. (3 de diciembre de 1993). «The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14». Cell 75 (5): 843-854. ISSN 0092-8674. PMID 8252621. doi:10.1016/0092-8674(93)90529-y. Consultado el 23 de agosto de 2024.
- ↑ Ruvkun, G. (26 de octubre de 2001). «Molecular biology. Glimpses of a tiny RNA world.». Science 294 (5543): 797-9. PMID 11679654. doi:10.1126/science.1066315.
- ↑ Glasser V. Tapping miRNA-Regulated Pathways. Genetic Eng Biotech News Mar 1 2008 (Vol. 28, No. 5) [1]
- ↑ a b c Saumet A, Lecellier CH (2006). «Anti-viral RNA silencing: do we look like plants?». Retrovirology 3 (3): 3. PMID 16409629. doi:10.1186/1742-4690-3-3.
- ↑ a b c Bartel, David P. (23 de enero de 2004). «MicroRNAs: genomics, biogenesis, mechanism, and function». Cell 116 (2): 281-297. ISSN 0092-8674. PMID 14744438. doi:10.1016/s0092-8674(04)00045-5. Consultado el 23 de agosto de 2024.
- ↑ Bentwich, Isaac; Avniel, Amir; Karov, Yael; Aharonov, Ranit; Gilad, Shlomit; Barad, Omer; Barzilai, Adi; Einat, Paz et al. (Julio de 2005). «Identification of hundreds of conserved and nonconserved human microRNAs». Nature Genetics 37 (7): 766-770. ISSN 1061-4036. PMID 15965474. doi:10.1038/ng1590. Consultado el 23 de agosto de 2024.
- ↑ Wightman, B.; Ha, I.; Ruvkun, G. (3 de diciembre de 1993). «Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans». Cell 75 (5): 855-862. ISSN 0092-8674. PMID 8252622. doi:10.1016/0092-8674(93)90530-4. Consultado el 23 de agosto de 2024.
- ↑ Ambros, Victor (13 de junio de 2003). «MicroRNA pathways in flies and worms: growth, death, fat, stress, and timing». Cell 113 (6): 673-676. ISSN 0092-8674. PMID 12809598. doi:10.1016/s0092-8674(03)00428-8. Consultado el 23 de agosto de 2024.
- ↑ Bartel, David P.; Chen, Chang-Zheng (Mayo de 2004). «Micromanagers of gene expression: the potentially widespread influence of metazoan microRNAs». Nature Reviews. Genetics 5 (5): 396-400. ISSN 1471-0056. PMID 15143321. doi:10.1038/nrg1328. Consultado el 23 de agosto de 2024.
- ↑ Lu, Cheng; Tej, Shivakundan Singh; Luo, Shujun; Haudenschild, Christian D.; Meyers, Blake C.; Green, Pamela J. (2 de septiembre de 2005). «Elucidation of the small RNA component of the transcriptome». Science (New York, N.Y.) 309 (5740): 1567-1569. ISSN 1095-9203. PMID 16141074. doi:10.1126/science.1114112. Consultado el 23 de agosto de 2024.
- ↑ He, Lin; Thomson, J. Michael; Hemann, Michael T.; Hernando-Monge, Eva; Mu, David; Goodson, Summer; Powers, Scott; Cordon-Cardo, Carlos et al. (9 de junio de 2005). «A microRNA polycistron as a potential human oncogene». Nature 435 (7043): 828-833. ISSN 1476-4687. PMC 4599349. PMID 15944707. doi:10.1038/nature03552. Consultado el 23 de agosto de 2024.
- ↑ Kim, V. Narry (Mayo de 2005). «MicroRNA biogenesis: coordinated cropping and dicing». Nature Reviews. Molecular Cell Biology 6 (5): 376-385. ISSN 1471-0072. PMID 15852042. doi:10.1038/nrm1644. Consultado el 23 de agosto de 2024.
- ↑ Zhao, Yong; Srivastava, Deepak (Abril de 2007). «A developmental view of microRNA function». Trends in Biochemical Sciences 32 (4): 189-197. ISSN 0968-0004. PMID 17350266. doi:10.1016/j.tibs.2007.02.006. Consultado el 23 de agosto de 2024.
- ↑ Zhao, Yong; Samal, Eva; Srivastava, Deepak (14 de julio de 2005). «Serum response factor regulates a muscle-specific microRNA that targets Hand2 during cardiogenesis». Nature 436 (7048): 214-220. ISSN 1476-4687. PMID 15951802. doi:10.1038/nature03817. Consultado el 23 de agosto de 2024.
- ↑ O'Donnell, Kathryn A.; Wentzel, Erik A.; Zeller, Karen I.; Dang, Chi V.; Mendell, Joshua T. (Junio de 2005). «c-Myc-regulated microRNAs modulate E2F1 expression». Nature (en inglés) 435 (7043): 839-843. ISSN 1476-4687. doi:10.1038/nature03677. Consultado el 23 de agosto de 2024.
- ↑ a b «DROSHA drosha ribonuclease III [Homo sapiens (human)] - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 23 de agosto de 2024.
- ↑ «pasha partner of drosha [Drosophila melanogaster (fruit fly)] - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 23 de agosto de 2024.
- ↑ Denli, Ahmet M.; Tops, Bastiaan B. J.; Plasterk, Ronald H. A.; Ketting, René F.; Hannon, Gregory J. (11 de noviembre de 2004). «Processing of primary microRNAs by the Microprocessor complex». Nature 432 (7014): 231-235. ISSN 1476-4687. PMID 15531879. doi:10.1038/nature03049. Consultado el 23 de agosto de 2024.
- ↑ Bernstein, E.; Caudy, A. A.; Hammond, S. M.; Hannon, G. J. (18 de enero de 2001). «Role for a bidentate ribonuclease in the initiation step of RNA interference». Nature 409 (6818): 363-366. ISSN 0028-0836. PMID 11201747. doi:10.1038/35053110. Consultado el 23 de agosto de 2024.
- ↑ Kurihara, Yukio; Watanabe, Yuichiro (24 de agosto de 2004). «Arabidopsis micro-RNA biogenesis through Dicer-like 1 protein functions». Proceedings of the National Academy of Sciences of the United States of America 101 (34): 12753-12758. ISSN 0027-8424. PMID 15314213. doi:10.1073/pnas.0403115101. Consultado el 23 de agosto de 2024.
- ↑ Gao, Fen-Biao (Enero de 2008). «Posttranscriptional control of neuronal development by microRNA networks». Trends in Neurosciences 31 (1): 20-26. ISSN 0166-2236. PMC 3545480. PMID 18054394. doi:10.1016/j.tins.2007.10.004. Consultado el 23 de agosto de 2024.
- ↑ Stark, Alexander; Bushati, Natascha; Jan, Calvin H.; Kheradpour, Pouya; Hodges, Emily; Brennecke, Julius; Bartel, David P.; Cohen, Stephen M. et al. (1 de enero de 2008). «A single Hox locus in Drosophila produces functional microRNAs from opposite DNA strands». Genes & Development 22 (1): 8-13. ISSN 0890-9369. PMC 2151017. PMID 18172160. doi:10.1101/gad.1613108. Consultado el 23 de agosto de 2024.
- ↑ Filipowicz, Witold; Jaskiewicz, Lukasz; Kolb, Fabrice A.; Pillai, Ramesh S. (Junio de 2005). «Post-transcriptional gene silencing by siRNAs and miRNAs». Current Opinion in Structural Biology 15 (3): 331-341. ISSN 0959-440X. PMID 15925505. doi:10.1016/j.sbi.2005.05.006. Consultado el 23 de agosto de 2024.
- ↑ MacRae, Ian J.; Doudna, Jennifer A. (Febrero de 2007). «Ribonuclease revisited: structural insights into ribonuclease III family enzymes». Current Opinion in Structural Biology 17 (1): 138-145. ISSN 0959-440X. PMID 17194582. doi:10.1016/j.sbi.2006.12.002. Consultado el 23 de agosto de 2024.
- ↑ «DGCR8 DGCR8 microprocessor complex subunit [Homo sapiens (human)] - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 23 de agosto de 2024.
- ↑ Zeng, Yan; Cullen, Bryan R. (29 de julio de 2005). «Efficient processing of primary microRNA hairpins by Drosha requires flanking nonstructured RNA sequences». The Journal of Biological Chemistry 280 (30): 27595-27603. ISSN 0021-9258. PMID 15932881. doi:10.1074/jbc.M504714200. Consultado el 23 de agosto de 2024.
- ↑ a b Han, Jinju; Lee, Yoontae; Yeom, Kyu-Hyeon; Nam, Jin-Wu; Heo, Inha; Rhee, Je-Keun; Sohn, Sun Young; Cho, Yunje et al. (2 de junio de 2006). «Molecular basis for the recognition of primary microRNAs by the Drosha-DGCR8 complex». Cell 125 (5): 887-901. ISSN 0092-8674. PMID 16751099. doi:10.1016/j.cell.2006.03.043. Consultado el 23 de agosto de 2024.
- ↑ Preall, Jonathan B.; He, Zhengying; Gorra, Jeffrey M.; Sontheimer, Erik J. (7 de marzo de 2006). «Short interfering RNA strand selection is independent of dsRNA processing polarity during RNAi in Drosophila». Current biology: CB 16 (5): 530-535. ISSN 0960-9822. PMID 16527750. doi:10.1016/j.cub.2006.01.061. Consultado el 23 de agosto de 2024.
- ↑ Gregory, Richard I.; Chendrimada, Thimmaiah P.; Cooch, Neil; Shiekhattar, Ramin (18 de noviembre de 2005). «Human RISC couples microRNA biogenesis and posttranscriptional gene silencing». Cell 123 (4): 631-640. ISSN 0092-8674. PMID 16271387. doi:10.1016/j.cell.2005.10.022. Consultado el 23 de agosto de 2024.
- ↑ Zhan, Mei; Miller, Chris P.; Papayannopoulou, Thalia; Stamatoyannopoulos, George; Song, Chao-Zhong (Julio de 2007). «MicroRNA expression dynamics during murine and human erythroid differentiation». Experimental Hematology 35 (7): 1015-1025. ISSN 0301-472X. PMC 1955220. PMID 17588470. doi:10.1016/j.exphem.2007.03.014. Consultado el 23 de agosto de 2024.
- ↑ Cifuentes, Daniel; Xue, Huiling; Taylor, David W.; Patnode, Heather; Mishima, Yuichiro; Cheloufi, Sihem; Ma, Enbo; Mane, Shrikant et al. (25 de junio de 2010). «A novel miRNA processing pathway independent of Dicer requires Argonaute2 catalytic activity». Science (New York, N.Y.) 328 (5986): 1694-1698. ISSN 1095-9203. PMC 3093307. PMID 20448148. doi:10.1126/science.1190809. Consultado el 23 de agosto de 2024.
- ↑ Lee, Heng-Chi; Li, Liande; Gu, Weifeng; Xue, Zhihong; Crosthwaite, Susan K.; Pertsemlidis, Alexander; Lewis, Zachary A.; Freitag, Michael et al. (25 de junio de 2010). «Diverse pathways generate microRNA-like RNAs and Dicer-independent small interfering RNAs in fungi». Molecular Cell 38 (6): 803-814. ISSN 1097-4164. PMC 2902691. PMID 20417140. doi:10.1016/j.molcel.2010.04.005. Consultado el 23 de agosto de 2024.
- ↑ Sen, George L.; Blau, Helen M. (Junio de 2005). «Argonaute 2/RISC resides in sites of mammalian mRNA decay known as cytoplasmic bodies». Nature Cell Biology 7 (6): 633-636. ISSN 1465-7392. PMID 15908945. doi:10.1038/ncb1265. Consultado el 23 de agosto de 2024.
- ↑ Lian, Shangli; Jakymiw, Andrew; Eystathioy, Theophany; Hamel, John C.; Fritzler, Marvin J.; Chan, Edward K. L. (Febrero de 2006). «GW bodies, microRNAs and the cell cycle». Cell Cycle (Georgetown, Tex.) 5 (3): 242-245. ISSN 1551-4005. PMID 16418578. doi:10.4161/cc.5.3.2410. Consultado el 23 de agosto de 2024.
- ↑ Jakymiw, Andrew; Lian, Shangli; Eystathioy, Theophany; Li, Songqing; Satoh, Minoru; Hamel, John C.; Fritzler, Marvin J.; Chan, Edward K. L. (Diciembre de 2005). «Disruption of GW bodies impairs mammalian RNA interference». Nature Cell Biology 7 (12): 1267-1274. ISSN 1465-7392. PMID 16284622. doi:10.1038/ncb1334. Consultado el 23 de agosto de 2024.
- ↑ Eulalio, Ana; Behm-Ansmant, Isabelle; Schweizer, Daniel; Izaurralde, Elisa (Junio de 2007). «P-body formation is a consequence, not the cause, of RNA-mediated gene silencing». Molecular and Cellular Biology 27 (11): 3970-3981. ISSN 0270-7306. PMC 1900022. PMID 17403906. doi:10.1128/MCB.00128-07. Consultado el 23 de agosto de 2024.
- ↑ a b Eulalio, Ana; Huntzinger, Eric; Izaurralde, Elisa (11 de enero de 2008). «Getting to the root of miRNA-mediated gene silencing». Cell 132 (1): 9-14. ISSN 0092-8674. PMID 18191211. doi:10.1016/j.cell.2007.12.024. Consultado el 23 de agosto de 2024.
- ↑ Eulalio, Ana; Rehwinkel, Jan; Stricker, Mona; Huntzinger, Eric; Yang, Schu-Fee; Doerks, Tobias; Dorner, Silke; Bork, Peer et al. (15 de octubre de 2007). «Target-specific requirements for enhancers of decapping in miRNA-mediated gene silencing». Genes & Development 21 (20): 2558-2570. ISSN 0890-9369. PMC 2000321. PMID 17901217. doi:10.1101/gad.443107. Consultado el 23 de agosto de 2024.
- ↑ Kozak, Marilyn (1 de noviembre de 2008). «Faulty old ideas about translational regulation paved the way for current confusion about how microRNAs function». Gene 423 (2): 108-115. ISSN 0378-1119. PMID 18692553. doi:10.1016/j.gene.2008.07.013. Consultado el 23 de agosto de 2024.
- ↑ Vasudevan, Shobha; Tong, Yingchun; Steitz, Joan A. (21 de diciembre de 2007). «Switching from repression to activation: microRNAs can up-regulate translation». Science (New York, N.Y.) 318 (5858): 1931-1934. ISSN 1095-9203. PMID 18048652. doi:10.1126/science.1149460. Consultado el 23 de agosto de 2024.
- ↑ Check Hayden, Erika (31 de julio de 2008). «Thousands of proteins affected by miRNAs». Nature 454 (7204): 562. ISSN 1476-4687. PMID 18668066. doi:10.1038/454562b. Consultado el 23 de agosto de 2024.
- ↑ Selbach, Matthias; Schwanhäusser, Björn; Thierfelder, Nadine; Fang, Zhuo; Khanin, Raya; Rajewsky, Nikolaus (4 de septiembre de 2008). «Widespread changes in protein synthesis induced by microRNAs». Nature 455 (7209): 58-63. ISSN 1476-4687. PMID 18668040. doi:10.1038/nature07228. Consultado el 23 de agosto de 2024.
- ↑ Baek, Daehyun; Villén, Judit; Shin, Chanseok; Camargo, Fernando D.; Gygi, Steven P.; Bartel, David P. (4 de septiembre de 2008). «The impact of microRNAs on protein output». Nature 455 (7209): 64-71. ISSN 1476-4687. PMC 2745094. PMID 18668037. doi:10.1038/nature07242. Consultado el 23 de agosto de 2024.
- ↑ Lewis, Benjamin P.; Burge, Christopher B.; Bartel, David P. (14 de enero de 2005). «Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets». Cell 120 (1): 15-20. ISSN 0092-8674. PMID 15652477. doi:10.1016/j.cell.2004.12.035. Consultado el 23 de agosto de 2024.
- ↑ Shan, Ge; Li, Yujing; Zhang, Junliang; Li, Wendi; Szulwach, Keith E.; Duan, Ranhui; Faghihi, Mohammad A.; Khalil, Ahmad M. et al. (Agosto de 2008). «A small molecule enhances RNA interference and promotes microRNA processing». Nature Biotechnology 26 (8): 933-940. ISSN 1546-1696. PMC 2831467. PMID 18641635. doi:10.1038/nbt.1481. Consultado el 23 de agosto de 2024.
- ↑ Lecellier, Charles-Henri; Dunoyer, Patrice; Arar, Khalil; Lehmann-Che, Jacqueline; Eyquem, Stephanie; Himber, Christophe; Saïb, Ali; Voinnet, Olivier (22 de abril de 2005). «A cellular microRNA mediates antiviral defense in human cells». Science (New York, N.Y.) 308 (5721): 557-560. ISSN 1095-9203. PMID 15845854. doi:10.1126/science.1108784. Consultado el 23 de agosto de 2024.
- ↑ Jopling, Catherine L.; Yi, Minkyung; Lancaster, Alissa M.; Lemon, Stanley M.; Sarnow, Peter (2 de septiembre de 2005). «Modulation of hepatitis C virus RNA abundance by a liver-specific MicroRNA». Science (New York, N.Y.) 309 (5740): 1577-1581. ISSN 1095-9203. PMID 16141076. doi:10.1126/science.1113329. Consultado el 23 de agosto de 2024.
- ↑ a b Lodish, Harvey F.; Zhou, Beiyan; Liu, Gwen; Chen, Chang-Zheng (Febrero de 2008). «Micromanagement of the immune system by microRNAs». Nature Reviews. Immunology 8 (2): 120-130. ISSN 1474-1741. PMID 18204468. doi:10.1038/nri2252. Consultado el 23 de agosto de 2024.
- ↑ Esquela-Kerscher, Aurora; Slack, Frank J. (Abril de 2006). «Oncomirs - microRNAs with a role in cancer». Nature Reviews. Cancer 6 (4): 259-269. ISSN 1474-175X. PMID 16557279. doi:10.1038/nrc1840. Consultado el 23 de agosto de 2024.
- ↑ Calin, George Adrian; Sevignani, Cinzia; Dumitru, Calin Dan; Hyslop, Terry; Noch, Evan; Yendamuri, Sai; Shimizu, Masayoshi; Rattan, Sashi et al. (2 de marzo de 2004). «Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers». Proceedings of the National Academy of Sciences of the United States of America 101 (9): 2999-3004. ISSN 0027-8424. PMID 14973191. doi:10.1073/pnas.0307323101. Consultado el 23 de agosto de 2024.
- ↑ Sonoki, T.; Iwanaga, E.; Mitsuya, H.; Asou, N. (Noviembre de 2005). «Insertion of microRNA-125b-1, a human homologue of lin-4, into a rearranged immunoglobulin heavy chain gene locus in a patient with precursor B-cell acute lymphoblastic leukemia». Leukemia 19 (11): 2009-2010. ISSN 0887-6924. PMID 16151463. doi:10.1038/sj.leu.2403938. Consultado el 23 de agosto de 2024.
- ↑ Calin, George Adrian; Dumitru, Calin Dan; Shimizu, Masayoshi; Bichi, Roberta; Zupo, Simona; Noch, Evan; Aldler, Hansjuerg; Rattan, Sashi et al. (26 de noviembre de 2002). «Frequent deletions and down-regulation of micro- RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia». Proceedings of the National Academy of Sciences of the United States of America 99 (24): 15524-15529. ISSN 0027-8424. PMID 12434020. doi:10.1073/pnas.242606799. Consultado el 23 de agosto de 2024.
- ↑ Büssing, Ingo; Slack, Frank J.; Grosshans, Helge (Septiembre de 2008). «let-7 microRNAs in development, stem cells and cancer». Trends in Molecular Medicine 14 (9): 400-409. ISSN 1471-4914. PMID 18674967. doi:10.1016/j.molmed.2008.07.001. Consultado el 23 de agosto de 2024.
- ↑ Ma, Li; Weinberg, Robert A. (Septiembre de 2008). «Micromanagers of malignancy: role of microRNAs in regulating metastasis». Trends in genetics: TIG 24 (9): 448-456. ISSN 0168-9525. PMID 18674843. doi:10.1016/j.tig.2008.06.004. Consultado el 23 de agosto de 2024.
- ↑ Lau, N. C.; Lim, L. P.; Weinstein, E. G.; Bartel, D. P. (26 de octubre de 2001). «An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans». Science (New York, N.Y.) 294 (5543): 858-862. ISSN 0036-8075. PMID 11679671. doi:10.1126/science.1065062. Consultado el 23 de agosto de 2024.
- ↑ «lin-4 (gene) - WormBase : Nematode Information Resource». wormbase.org. Consultado el 23 de agosto de 2024.
- ↑ «let-7 (gene) - WormBase : Nematode Information Resource». wormbase.org. Consultado el 23 de agosto de 2024.
- ↑ Reinhart, B. J.; Slack, F. J.; Basson, M.; Pasquinelli, A. E.; Bettinger, J. C.; Rougvie, A. E.; Horvitz, H. R.; Ruvkun, G. (24 de febrero de 2000). «The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans». Nature 403 (6772): 901-906. ISSN 0028-0836. PMID 10706289. doi:10.1038/35002607. Consultado el 23 de agosto de 2024.
- ↑ «lin-14 (gene) - WormBase : Nematode Information Resource». wormbase.org. Consultado el 23 de agosto de 2024.
- ↑ «lin-28 (gene) - WormBase : Nematode Information Resource». wormbase.org. Consultado el 23 de agosto de 2024.
- ↑ «lin-41 (gene) - WormBase : Nematode Information Resource». wormbase.org. Consultado el 23 de agosto de 2024.
- ↑ «lin-29 (gene) - WormBase : Nematode Information Resource». wormbase.org. Consultado el 23 de agosto de 2024.
- ↑ «SRF serum response factor [Homo sapiens (human)] - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 23 de agosto de 2024.
- ↑ «miRBase entry: mmu-mir-1a-1». www.mirbase.org. Consultado el 23 de agosto de 2024.
- ↑ «miRBase entry: mmu-mir-1a-2». www.mirbase.org. Consultado el 23 de agosto de 2024.
- ↑ «HAND2 heart and neural crest derivatives expressed 2 [Homo sapiens (human)] - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 23 de agosto de 2024.
- ↑ «NOTCH1 notch receptor 1 [Homo sapiens (human)] - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 23 de agosto de 2024.
- ↑ Zhao, Yong; Srivastava, Deepak (Abril de 2007). «A developmental view of microRNA function». Trends in Biochemical Sciences 32 (4): 189-197. ISSN 0968-0004. PMID 17350266. doi:10.1016/j.tibs.2007.02.006. Consultado el 23 de agosto de 2024.
- ↑ «miRBase entry: mmu-mir-133a-1». www.mirbase.org. Consultado el 23 de agosto de 2024.
- ↑ «miRBase entry: mmu-mir-133a-2». www.mirbase.org. Consultado el 23 de agosto de 2024.
- ↑ «MEF2A myocyte enhancer factor 2A [Homo sapiens (human)] - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 23 de agosto de 2024.
- ↑ «MYOD1 myogenic differentiation 1 [Homo sapiens (human)] - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 23 de agosto de 2024.
- ↑ «HDAC4 histone deacetylase 4 [Homo sapiens (human)] - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 23 de agosto de 2024.
- ↑ «miRBase entry: mmu-mir-181a-2». www.mirbase.org. Consultado el 23 de agosto de 2024.
- ↑ «miRBase entry: mmu-mir-206». www.mirbase.org. Consultado el 23 de agosto de 2024.
- ↑ «miRBase entry: mmu-mir-126a». www.mirbase.org. Consultado el 23 de agosto de 2024.
- ↑ «miRBase entry: mmu-mir-200a». www.mirbase.org. Consultado el 23 de agosto de 2024.
- ↑ «miRBase entry: mmu-mir-30c-1». www.mirbase.org. Consultado el 23 de agosto de 2024.
- ↑ Wienholds, Erno; Kloosterman, Wigard P.; Miska, Eric; Alvarez-Saavedra, Ezequiel; Berezikov, Eugene; de Bruijn, Ewart; Horvitz, H. Robert; Kauppinen, Sakari et al. (8 de julio de 2005). «MicroRNA expression in zebrafish embryonic development». Science (New York, N.Y.) 309 (5732): 310-311. ISSN 1095-9203. PMID 15919954. doi:10.1126/science.1114519. Consultado el 23 de agosto de 2024.
- ↑ Murchison, Elizabeth P.; Stein, Paula; Xuan, Zhenyu; Pan, Hua; Zhang, Michael Q.; Schultz, Richard M.; Hannon, Gregory J. (15 de marzo de 2007). «Critical roles for Dicer in the female germline». Genes & Development 21 (6): 682-693. ISSN 0890-9369. PMC 1820942. PMID 17369401. doi:10.1101/gad.1521307. Consultado el 23 de agosto de 2024.
- ↑ Tang, Fuchou; Kaneda, Masahiro; O'Carroll, Dónal; Hajkova, Petra; Barton, Sheila C.; Sun, Y. Andrew; Lee, Caroline; Tarakhovsky, Alexander et al. (15 de marzo de 2007). «Maternal microRNAs are essential for mouse zygotic development». Genes & Development 21 (6): 644-648. ISSN 0890-9369. PMC 1820938. PMID 17369397. doi:10.1101/gad.418707. Consultado el 23 de agosto de 2024.