Leviviricetes
Leviviricetes es una clase de virus que infectan procariotas (bacterias y arqueas).[1] Contienen un genoma ARN monocatenario positivo y por lo tanto se incluyen en el Grupo IV de la Clasificación de Baltimore. Se caracterizan por una cápside carente de envoltura viral y estructuralmente definida por una simetría icosaédrica regular.[2]
| Leviviricetes | ||
|---|---|---|
 | ||
| Taxonomía | ||
| Dominio: | Riboviria | |
| Reino: | Orthornavirae | |
| Filo: | Lenarviricota | |
| Clase: | Leviviricetes | |
| Clasificación de Baltimore | ||
| Grupo: | IV (Virus ARN monocatenario positivo) | |
| Órdenes | ||
Esta clase incluye la gran mayoría de virus de ARN procariotas descritos. Antiguamente se clasificaban como una única familia Leviviridae que infectaba bacterias, pero la metagenómica y nuevos aislamientos ha llevado a la conclusión de que la antigua familia era más expansiva, ampliando la diversidad de levivirus que pueden estar infectado cualquier tipo de procariota.[3][4][5][6]
Clasificación
editarLa taxonomía establecida por el ICTV en 2021 es la siguiente:[1]
- Clase Leviviricetes
- Orden Norzivirales
- Familia Atkinsviridae
- Familia Duinviridae
- Familia Fiersviridae
- Familia Solspiviridae
- Orden Timlovirales
- Familia Blumeviridae
- Familia Steitzviridae
- Género Chimpevirus
- Género Hohglivirus
- Género Mahrahovirus
- Género Meihzavirus
- Género Nicedsevirus
- Género Sculuvirus
- Género Skrubnovirus
- Género Tetipavirus
- Género Winunavirus
- Orden Norzivirales
Morfología y estructura
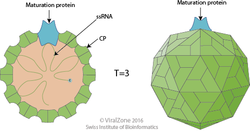
editarSon virus isométricos, muy pequeños que miden entre 20 y 27 nm siendo a su vez los virus de ARN más pequeños conocidos. Su cápside tiene 180 subunidades de un único tipo de proteína. Portan, una proteína de maduración, indispensable para la correcta formación de la cápside y el ensamblaje con el ARN, y también muy importante para el reconocimiento del hospedador.
Genoma
editarEl genoma es muy pequeño (3-4 kilobases) siendo el más pequeño entre los virus de ARN, y sólo tiene 4 genes: proteína de maduración (Prot. A), proteína de la cápside, la ARN replicasa (ARN polimerasa dependiente de ARN) y proteína de lisis.
Porta, además, secuencias específicas para el reconocimiento por parte de la ARN replicasa. La proteína de lisis requiere un cambio en la fase de lectura respecto de las otras (salto de +1 base), lo que le permite tener la información solapada con los otros genes.
El genoma, además, cuenta con zonas de complementariedad interna, lo que hace que se encuentre plegado. Los extremos 3’ y 5’ están protegidos por asociación a la proteína de maduración.
Ciclo de infección
editarLa proteína A reconoce la célula hospedadora a través de sus pili. El virus se une a ellos y se induce la entrada en la célula del ARN asociado a la proteína A. Una vez dentro, el ARN es leído como un mensajero por los ribosomas celulares que van a traducir su información.
Ahora bien, el virus va a necesitar que la información se traduzca en cantidades distintas. Será necesaria mucha traducción de la proteína estructural, pero menos del resto. Se usa un mecanismo de regulación que tiene que ver con la estructura terciaria del ARN.
Al principio, la región que codifica la proteína A está bloqueada, de modo que ésta no se sintetiza.
Sí se sintetiza, por el contrario, la proteína estructural. Conforme los ribosomas avanzan leyendo esta región, se va deshaciendo la estructura sterciaria, facilitando su traducción, y liberando el sitio de inicio de la traducción del gen de la ARN replicasa.
Se están traduciendo la proteína estructural y la ARN replicasa, pero como se necesita menos ARN replicasa, en cuanto hay suficiente proteína estructural, se asocian varios monómeros y bloquean la traducción de la ARN replicasa.
La ARN replicasa ejerce su función, que es replicar la cadena de ARN (+) a una de ARN (-), y a partir de esta, replicar nuevas copias de la cadena de ARN (+), que pueden funcionar como mensajeros o comenzar a encapsidarse.
La proteína de maduración se traduce durante el proceso de replicación de la hebra – a una +. Hay un momento en el proceso en el que la cadena naciente, tiene libre el sitio de inicio de la traducción, al que se une un ribosoma.
Ensamblaje y lisis
editarEl ensamblaje se produce de forma automática. La célula se lisa y se liberan los viriones. El proceso de replicación es muy efectivo, se forman muchos viriones.
Referencias
editar- ↑ a b «Virus Taxonomy: 2020 Release» (html). International Committee on Taxonomy of Viruses (ICTV) (en inglés). October 2018. Consultado el 13 octobre 2019.
- ↑ Eugene Koonin, Valerian V Doljja (2014). Virus World as an Evolutionary Network of Viruses and Capsidless Selfish Elements. Microbiology and Molecular Biology Reviews.
- ↑ Callanan J, Stockdale SR, Adriaenssens EM, Kuhn JH (January 2021). «Rename one class (Leviviricetes - formerly Allassoviricetes), rename one order (Norzivirales - formerly Levivirales), create one new order (Timlovirales), and expand the class to a total of six families, 420 genera and 883 species.». ResearchGate. doi:10.13140/RG.2.2.25363.40481.
- ↑ Leviviricetes Study Group Archivado el 13 de mayo de 2021 en Wayback Machine.. ICTV.
- ↑ Siddharth R. Krishnamurthy, Andrew B. Janowski,Guoyan Zhao, Dan Barouch, David Wang (2016). Hyperexpansion of RNA Bacteriophage Diversity. PlosOne.
- ↑ Yuri I. Wolf, Sukrit Silas, Yongjie Wang, Shuang Wu, Michael Bocek, Darius Kazlauskas, Mart Krupovic, Andrew Fire, Valerian V. Dolja & Eugene V. Koonin (2020). Doubling of the known set of RNA viruses by metagenomic analysis of an aquatic virome. Nature.
- Prescott, L.M. (199). Microbiología. McGraw-Hill Interamericana de España, S.A.U. ISBN 84-486-0261-7.